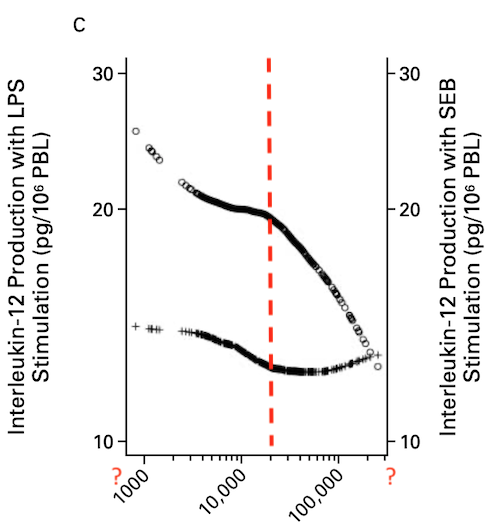
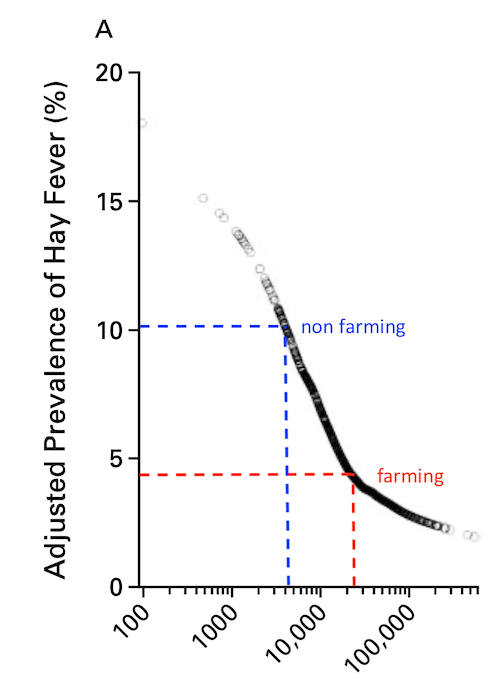
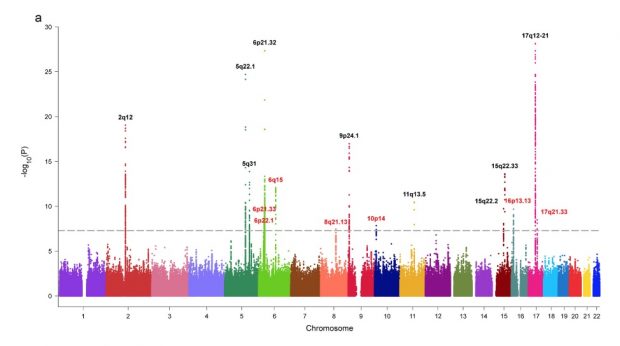
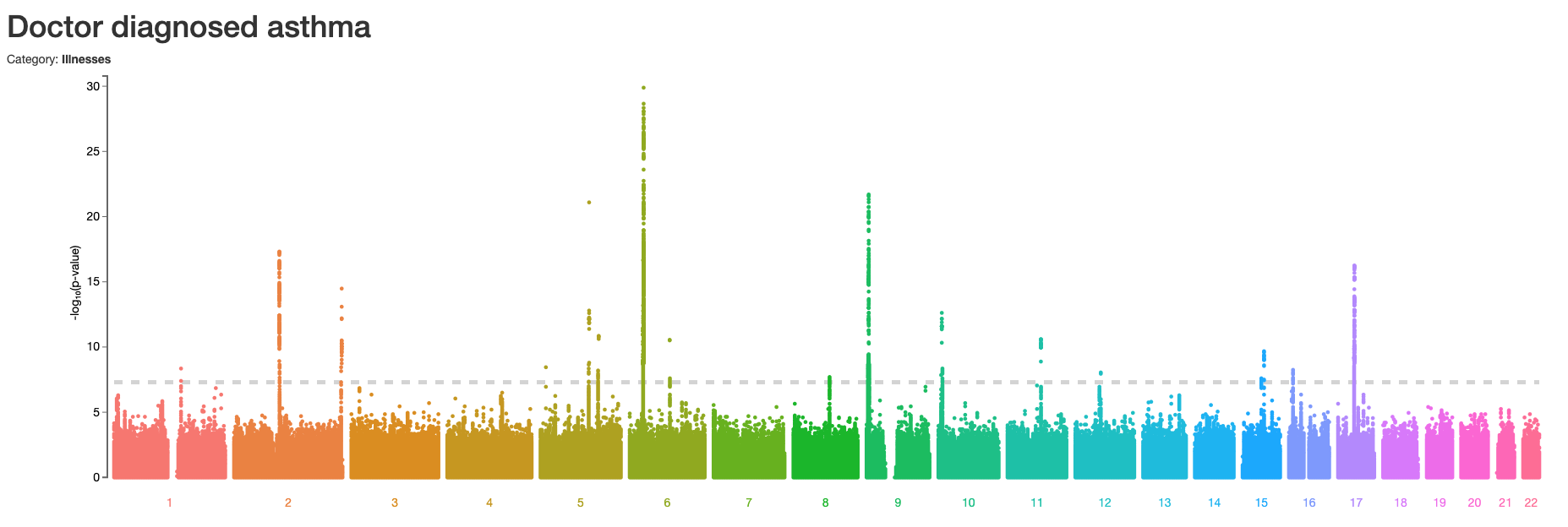
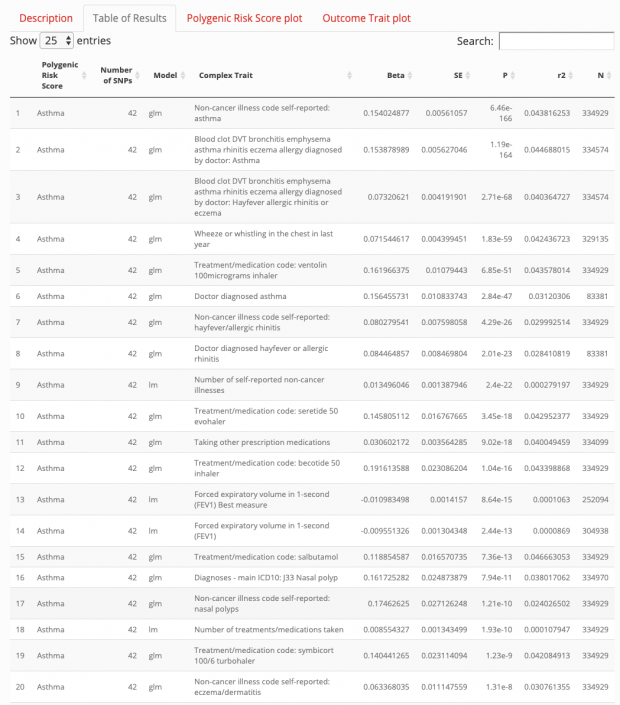
A recent Nature study showed Ebola reactivation in a previously infected patient:
The 2021 lineage shows considerably lower divergence than would be expected during sustained human-to-human transmission, which suggests a persistent infection with reduced replication or a period of latency.
The most recent viral genome shared 10 substitutions that evolved during the previous epidemic making it unlikely that there was a new animal spillover event. I always wondered how second & third wave of COVID-19 started in Germany. Was it really a new spread or just a reactivation? A Frontiers review concluded from the existing literature that
our study, consisting more than a total of 113,715 patients, indicates that the RP-SARS-CoV-2 scenario occurs plausibly due to reactivation, reinfection, viral shedding, or testing errors.
So far, there are 240 documented COVID-19 cases of reinfection reported worldwide according to the reinfection tracker. In the case of Ebola there is a known viral persistence in semen while transmission through milk and cervicovaginal fluid is also possible (similar for COVID-19 although neuronal persistence seems to be more relevant). Maybe we need more immunological studies particular in long COVID if there is a continuous or intermittent antigenic stimulation due to persistence of an antigenic reservoir.
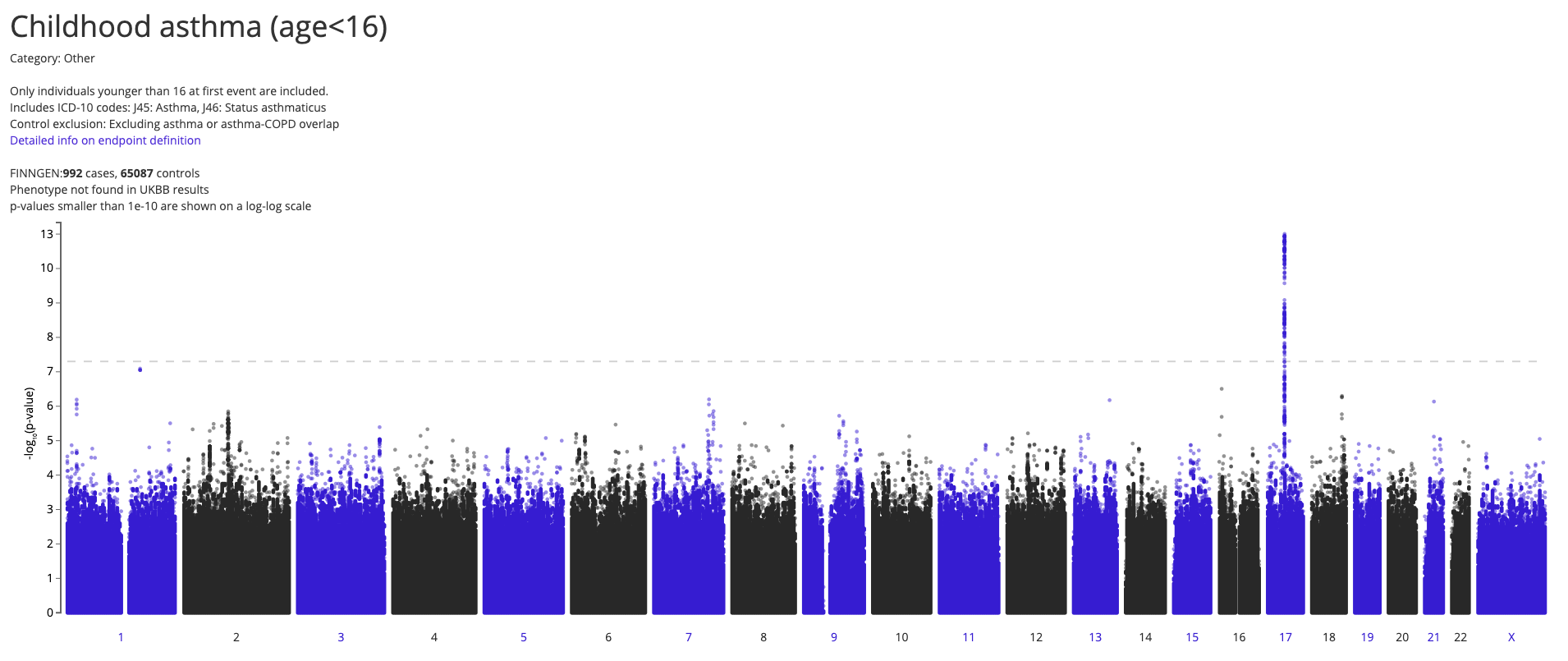
The fourth wave in Germany is caused by the delta variant, reactivation of alpha is certainly not a major factor. So we will only know in the next few years if reactivation is responsible for small regional outbreaks in unvaccinated communities.